Sequenzierung der nächsten Generation (NGS) zur hypothesenfreien genomischen Erregerdetektion in potenziell polymikrobiell kontaminiertem Probenmaterial – Evaluation am Beispiel formalifixierter, -paraffinierter Gewebeproben von Patienten mit invasiven Mykosen1
Aus dem Fachbereich Tropenmedizin am Bernhard-Nocht-Institut (Leiterin: Oberfeldarzt Dr. D. Wiemer) des Bundeswehrkrankenhauses Hamburg (Chefarzt: Generalarzt Dr. J. Hoitz)
Hintergrund: Wenn bei unspezifischem Verdacht auf eine invasive bakterielle oder fungale Infektion aus primär steril gewonnenem Probenmaterial ein molekularer Erregernachweis angestrebt wird, werden von vielen Instituten inzwischen panbakterielle bzw. panfungale Polymerasekettenreaktionen (PCR) mit anschließender Sanger-Sequenzierung zur Erregeridentifikation trotz eingeschränkter Standardisierbarkeit diagnostisch angeboten.
Bei polymikrobiellen Infektionen oder primär mikrobiell kontaminiertem Probenmaterial sind die Resultate solcher Untersuchungen jedoch häufig nicht auswertbar, so dass als neuer diagnostischer Ansatz für die hypothesenfreie molekulare Erregerdiagnostik die „Sequenzierung der nächsten Generation“ (Next-Generation Sequencing, NGS) zunehmend evaluiert wird.
Material und Methoden: In dieser Proof-of-Principle-Untersuchung wurde ein Panel aus 17 histologisch charakterisierten, formalinfixierten, paraffinierten Proben von Patienten mit seltenen und tropischen invasiven Mykosen mit panfungaler PCR und anschließender Sanger-Sequenzierung sowie mittels NGS untersucht.
Ergebnisse: Erwartungsgemäß erwiesen sich die nichtsteril gelagerten histologischen Proben als polymikrobiell kontaminiert. Entsprechend gelang ein mit der Histologie übereinstimmender Erregernachweis mit panfungaler PCR und Sanger-Sequenzierung nur in 2 von 17 Fällen. Mittels NGS mit quan-ti-ta-tiver Wichtung der nachgewiesenen Erreger--Nukleinsäure konnte dieser Anteil auf 5 von 17 Fällen gesteigert werden.
Diskussion: Mit NGS steht ein neues diagnostisches Tool zur Verfügung, das geeignet ist, auch bei diagnostisch anspruchsvollem Probenmaterial eine hypothesenfreie Erregerdiagnostik zu ermöglichen. Zu berücksichtigen sind Sensitivitätsgrenzen und eine teils schwierige Interpretierbarkeit bei polymikrobiell kontaminiertem Probenmaterial. Die hochkomplexe bioinformatische Auswertung bedarf weiterer Standardisierung. Mittelfristig kann die diagnostische NGS dazu beizutragen, bei komplexen infektiologischen Fällen im Inland wie auch im Einsatz die zuverlässige Identifikation des ätiologischen relevanten infektiösen Agens zu unterstützen.
Schlüsselwörter: NGS; Sequenzierung der nächsten Generation; hypothesenfreie Infektionsdiagnostik; invasive Mykosen; formalinfixierte, paraffinierte Proben; molekulare Diagnostik; tropische Infektionserreger
Keywords: NGS; next generation sequencing; unbiased detection of pathogens; formalin-fixed paraffin-embedded; molecular diagnostics; tropical infectious agents
Hintergrund
Zuverlässige Ergebnisse mikrobiologischer Untersuchungen, insbesondere kultureller Art, setzen adäquate präanalytische Bedingungen voraus [1]. Können diese Voraussetzungen in Einsätzen mit stark dislozierten Kräften nicht erfüllt werden, muss mit einem Absterben ätiologisch relevanter Erreger vor Eintreffen im diagnostischen Labor gerechnet werden.
Darüber hinaus kann die Inaktivierung von Infektionserregern die Diagnostik erschweren. Dies ist beispielsweise der Fall, wenn bei der initialen Probenentnahme die Möglichkeit einer infektiösen Ätiologie nicht berücksichtigt, das Probenmaterial entsprechend zur histopathologischen Aufarbeitung in 4 % gepuffertem Formalin zwecks Erhalts der Gewebsstruktur fixiert und anschließend im pathologischen Labor in Paraffin eingebettet wird. Liefert die Histologie nun Hinweise auf eine infektiöse Genese einer inflammatorischen Reaktion, so ist durch die formalinbedingte Erregerinaktivierung ein kultureller Nachweis nicht mehr erfolgversprechend. Auch die Sensitivität molekularer Diagnostikverfahren, z. B. der PCR, ist durch die Formalinfixierung aufgrund von Nukleinsäure- und Proteinvernetzung und die schwierige DNA-Extraktion aus dem paraffinierten Gewebe deutlich reduziert [2 - 5]. Wenn der mikroskopische Erregernachweis kein schlüssiges Ergebnis liefert, so ist der molekulare Nachweis aus formalinfixiertem Probenmaterial dennoch der erfolgversprechendste Ansatz, falls frisches Probenmaterial nicht oder nur unter erheblicher Belastung für den Patienten gewonnen werden kann [5]. Die Sensitivität des Erregernachweises wird dabei von Faktoren wie Probenalter und Erregerdichte beeinflusst [5]. Beste Ergebnisse können für PCRs erwartet werden, die sehr kurze Fragmente amplifizieren, da die formalinbedingte Quervernetzung von DNA-Strängen sowie Protein-DNA-Quervernetzungen die Amplifikation größerer Fragmente verhindern. Solche Vernetzungsereignisse sind – stochastisch verteilt – etwa alle 1 000 Basenpaare zu erwarten und reduzieren die Zuverlässigkeit von PCRs mit längeren Amplifikaten. Dies gilt insbesondere dann, wenn per se wenig Erreger-DNA in den Proben enthalten ist [5, 6].
Ein Nachteil gezielter PCR-Diagnostik besteht darin, dass die primergestützte Nukleinsäureamplifikation nur Erbinformation definierter Erreger oder Erregergruppen erfasst. Dies bedeutet für den Einsender, dass er zum Zeitpunkt der Diagnostikanforderung symptomabgeleitet klare Vorstellungen zur abzuklärenden Verdachtsdiagnose haben muss. Sind die Symptome des Patienten dagegen unspezifisch und können durch eine Vielzahl möglicher Erreger induziert werden, kann die umfassende und zugleich ökonomisch vernünftige Auswahl des einzusetzenden PCR-Panels eine differenzialdiagnostische Herausforderung darstellen. Entsprechend ist die Wirtschaftlichkeit des Einsatzes molekularer Erregerdiagnostik teils zweifelhaft [7], wenngleich entsprechende PCR-Techniken im Sanitätsdienst zum Einsatz kommen [8, 9].
Eine mögliche Alternative zu genus- oder speziesspezifischen PCR-Verfahren besteht in der Durchführung sogenannter panbakterieller oder panfungaler PCRs mit konsekutiver Sanger-Sequenzierung [10] zur sequenzgestützten Erregeridentifikation von Bakterien und Pilzen im Probenmaterial [11]. Zielstrukturen solcher PCRs sind DNA-Abschnitte, die für ribosomale RNA kodieren. Die Primer der PCRs binden an phylogenetisch hochkonservierte Regionen, die Identifikation erfolgt anhand hochvariabler Sequenzen dazwischen. Entsprechende Verfahren sind meist wenig standardisiert und somit – insbesondere bei negativem Reaktionsausfall – in ihrer diagnostischen Reliabilität zweifelhaft [11], können aber im positiven Fall wertvolle Hinweise liefern. Ein ergänzender diagnostischer Wert dieser Verfahren ist vor allem für steril gewonnenes Probenmaterial bei Infektionen in primär sterilen Kompartimenten, wie Endokarditiden [12], belegt. Voraussetzung für ein zuverlässiges Sequenzierergebnis ist jedoch die Anwendung an primär sterilem Probenmaterial, in dem sich nur der ätiologisch relevante Erreger in Reinkultur befindet. Bei Mischkulturen oder (poly-)mikrobiell kontaminierten Proben kommt es zu Sequenzüberlagerungen in der Sanger-Sequenzierung, die meist keine zuverlässige Erregeridentifizierung zulassen [13]. Mit ebensolchen mikrobiellen Kontaminationen muss bei formalinfixiertem, paraffiniertem Probenmaterial jedoch nahezu regelmäßig gerechnet werden – sei es durch die nichtsterile Lagerung der Paraffinblöckchen oder Kontaminationen im Paraffin selbst. Entsprechend ist die diagnostische Aussagekraft eingeschränkt.
Eine potenzielle Alternative bietet die diagnostische Anwendung von NGS am Primärmaterial. Dabei erfolgt eine unspezifische Sequenzierung der in der Probe enthaltenen DNA in Form kurzer Sequenzfragmente, sogenannter „Reads“, mit der anschließenden Option des bioinformatischen Abgleichs solcher Reads mit in Datenbanken hinterlegten Erregergenomen.
Auf diese Weise ist es bereits erfolgreich gelungen, ätiologisch unklare Infektionsereignisse spezifischen Erregern zuzuordnen [14]. Auch aus primär nicht-sterilen Materialien kann, z. B. auf der Basis der relativen Häufigkeit erregerspezifischer Nukleinsäuresequenzen oder der Pathogenität solcherart molekular nachgewiesener Erreger, die Zuordnung der ätiologischen Relevanz angesichts einer bestehenden klinischen Symptomatik erfolgen [15]. Im Sanitätsdienst der Bundeswehr sind/werden diagnostische NGS-Plattformen zur mikrobiellen Erregerdiagnostik derzeit am Institut für Mikrobiologie der Bundeswehr in München etabliert.
Die Anwendung von NGS aus formalinfixiertem, paraffiniertem Probenmaterial im Allgemeinen [16] und zwecks Erregernachweis und -typisierung aus solchem Material im Besonderen [17] ist Gegenstand aktueller Evaluationsstudien. Die hier vorgestellte Untersuchung beschäftigt sich exemplarisch mit dem NGS-gestützten Nachweis invasiver, überwiegend tropischer Mykosen aus histologischem Probenmaterial.
Material und Methoden
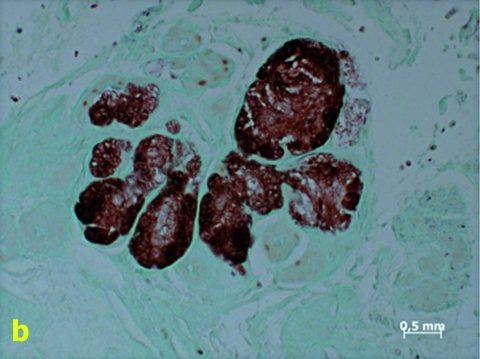
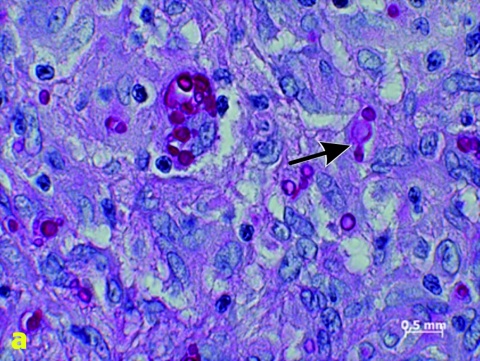
Hierzu wurden insgesamt siebzehn formalinfixierte paraffinierte Gewebeproben histologisch umfassend charakterisiert. Zu den histologischen Diagnosen gehörten die Chromoblastomykose (n = 3), die Coccidioidomykose (n = 2), die Histoplasmose (n = 3), die Histoplasmose oder Kryptokokkose bei histologisch schwieriger Diskriminierbarkeit (n = 1), die Mukormykose (n = 2), das Myzetom (n = 3) und die Rhinosporidiose (n = 2) (Abbildung 1).
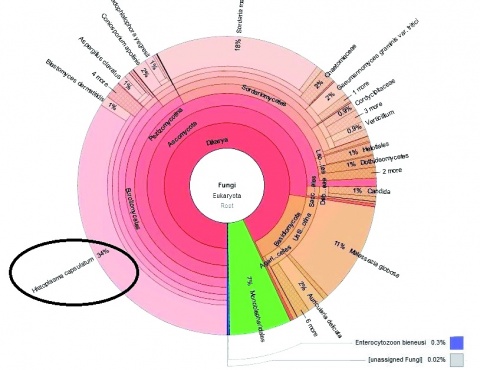
Anschließend wurden nach Extraktion der Nukleinsäure panfungale PCRs mit anschließender Sanger-Sequenzierung durchgeführt [18]. Ferner wurden die Proben mittels NGS (jeweils 1 Illumina v3-Lauf pro Probe) untersucht (Publikation in Vorbereitung). Um in der NGS-Analytik Kontamination von ätiologisch mutmaßlich relevanter Infektion zu diskriminieren, wurden in allen untersuchten Proben für die in Frage kommenden Erreger Mittelwert und Standardabweichung der Anzahl spezifischer Sequenzfragmente (Reads) bestimmt und verglichen.
Ergebnisse
Diskussion
Vom Stellenwert diagnostischer NGS – eine Bestandsaufnahme
Die NGS-Technologie bietet erstmals ein molekularbiologisches diagnostisches Verfahren, das eine Erregerdiagnostik auch im komplexen Probenmaterial ohne vorherigen konkreten Verdacht ermöglicht, sofern eine hinreichende Sequenziertiefe ermöglicht wird. Die technologischen Ansätze sind heterogen [10, 19 - 26] und teils noch im Stadium der Entwicklung bzw. Optimierung. Eine anschauliche Übersicht über den NGS-Ablauf in der Infektionsdiagnostik wurde von HASMANN et al. vorgestellt [27]. In früheren Arbeiten konnten Assoziationen zwischen infektiösen Krankheitserregern und Krankheiten unklarer Ätiologie erfolgreich belegt werden [14]. Ferner gelang der NGS-basierte Nachweis bakterieller Erreger aus zwei Dritteln untersuchter Urine in einer vorausgegangenen „Proof-of-Principle“-Untersuchung [27]. NGS eignet sich ferner auch für den Nachweis polymikrobiell verursachter Infektionen, wie am Beispiel der Anwendung an Probenmaterial aus Hirnabszessen erfolgreich belegt werden konnte [28]. Die zuverlässigsten diagnostischen Aussagen können mittels NGS aus primär sterilem Probenmaterial getroffen werden, wo bereits wenige Reads für eine Erregerdiagnostik genutzt werden können. So gelang WILSON et al. mittels NGS der Nachweis einer durch Leptospira santarosai verursachten Meningoenzephalitis anhand von nur 475 (aus mehr als drei Millionen) spezifischen Reads [29]. Erregeridentifikation basierend auf NGS-gestützter Analytik von im Probenmaterial vorhandener RNA (Ribonukleinsäure) ist ebenfalls möglich und erfasst z. B. RNA-Viren wie Influenza in respiratorischen Proben im sogenannten UMERS („unbiased nontargeted metagenomic RNA sequencing“) Ansatz [30].
Wenngleich die NGS-Technologie noch immer als kostspielig angesehen werden muss, fallen die Sequenzierkosten dramatisch. So konnten beispielsweise die Kosten für die Sequenzierung eines humanen Genoms von circa 100 000 Euro noch vor wenigen Jahren aufgrund des technologischen Fortschritts auf mittlerweile nur noch etwa 1 000 Euro abgesenkt werden [10]. Insbesondere die Einführung von kleinen Sequenzierautomaten (etwa in der Größe von Laserdruckern) macht die NGS-Technologie auch unter wehrmedizinischen Gesichtspunkten für die Diagnostik im Einsatz mittelfristig interessant. In einer früheren vergleichenden Evaluation dieser kleinen „Werkbank“-Sequenzierautomaten zeigte sich, dass insbesondere das auch in dieser Studie eingesetzte MiSeq®-System (Illumina) den Produkten 454 GS™ Junior (Roche, Basel, Schweiz) und Ion Torrent PGM™ (Life Technologies, Carlsbad, Kalifornien, USA) hinsichtlich der Seltenheit von Sequenzierfehlern überlegen ist [31].
Die bis dato recht anspruchsvolle und benutzerunfreundliche Aufbereitung der Sequenzierdaten stellt derzeit eine der Hauptlimitationen der breiten diagnostischen Anwendung der NGS-Technologie dar [32]. Hier bleibt die weitere Automatisierung und Standardisierung, die für diagnostische Routineabläufe unerlässlich ist, abzuwarten bzw. bedarfsgerecht aktiv mitzugestalten. Dies gilt auch für die Qualität zugrundeliegender Datenbanken.
Wenngleich die Anwendung von NGS an formalinfixiertem, paraffiniertem Gewebe keine Neuheit darstellt [16, 17], ist der NGS-basierte Nachweis ätiologisch relevanter Erreger aus solchen Materialien eine diagnostische Herausforderung. Nichterregerspezifische molekulare diagnostische Ansätze wie NGS können leicht durch Kontaminationen aufgrund von Mikroorganismen aus der Umwelt, die zum Beispiel im Paraffin eingegossen sind, beeinflusst werden. Diese Problematik wurde auch in der hier vorgestellten Studie sowohl hinsichtlich der panfungalen PCRs als auch des NGS-Ansatzes sehr deutlich. Die auf panfungale PCRs gestützte Sanger-Sequenzanalytik invasiver Mykosen aus Paraffingewebe erwies sich als kontaminationsanfällig und nur bei frischen, wenig inhibierten Proben als zuverlässig. Eine mögliche Alternative sind speziesspezifische PCRs [33 - 40], jedoch erfordert deren Auswahl einen diagnostischen Anfangsverdacht.
Schwierige Histologie bei invasiven Mykosen
Auch die klassische Histologie hilft beim Verdacht auf invasive Mykosen nicht immer weiter. Ihre Zuverlässigkeit wird durch eine Vielzahl von Faktoren beeinflusst, darunter eine kritische Mindestdichte von Erregern im untersuchten Gewebe sowie ein hohes Maß an Expertise des Untersuchers. Entsprechend legen vergleichende Untersuchungen zwischen Histologie und Kultur, wobei letztgenannte nicht aus formalinfixiertem Gewebe erfolgen kann, eine Übereinstimmung von nicht einmal 80 % nahe [41], so dass auch histologische Diagnosen mit Vorsicht zu interpretieren sind [35]. In diesem Fall erfolgte die histologische Begutachtung durch erfahrene Pathologen mit professionellem Bezug zu tropischen Infektionserkrankungen. Insbesondere mit Blick auf die Vielzahl von Gattungen und Spezies, die bei invasiven Mykosen ein histologisch ähnliches Bild bieten können, eignen sich invasive Mykosen besonders zur Demonstration der Vorzüge einer molekularen Diagnostik bei gegebener Interpretierbarkeit der Ergebnisse.
Nukleinsäureextraktion – ein oft unterschätzter erster Schritt
Eine erste wichtige Voraussetzung für die Zuverlässigkeit molekularer Diagnostikbefunde ist die Qualität der Nukleinsäureextraktion, die in der hier vorgestellten Untersuchung auch in über Jahre gelagerten Proben akzeptabel war. Alterdings wurden in einer Reihe von Proben auch Hinweise auf Inhibition beobachtet. Die vergleichende Testung alternativer Aufreinigungsverfahren [42] hätte zur weiteren Optimierung der Nukleinsäurepräparation beitragen können. Dies war jedoch aufgrund der geringen verfügbaren Menge an Probenmaterial nicht möglich, was eine unbestrittene Limitation der Studie darstellt.
Panfungale PCRs mit Sangersequenzierung – keine gute Option für kontaminierte Proben
Die eingesetzten panfungalen PCRs [42 - 47] waren unter standardisierten Bedingungen in Laboratorien von Industrieländern evaluiert und zum Teil auch für die Anwendung aus formalinfixiertem, paraffiniertem Gewebe für geeignet befunden worden. Die gilt insbesondere für den Nachweis der in Industrieländern gelegentlich nachgewiesenen invasiven Mykosen durch Candida spp. und Aspergillus spp. bei schwerst immunsupprimierten Patienten [42, 46, 47].
Da die Paraffinblöckchen mit den formalinfixierten Geweben über Jahre ohne besondere Schutzmaßnahmen gegen die Sedimentation von Pilzsporen gelagert waren, kann eine entsprechende Kontamination mit Umweltpilzsporen als hochwahrscheinlich angesehen werden. So sind die hohen Anteile an Kontaminationen mit Pilzen, denen keine humanpathogene Relevanz zugesprochen wird, zu erklären. Eine Alternativerklärung stellt die Sporenkontamination des Paraffins dar. Die panfungalen Primer reagierten in vielen Fällen bereitwilliger mit Umweltkontaminanten als mit dem tatsächlichen infektiösen Agens. Dieses Phänomen unterschiedlicher Bindungspräferenz der Primer ist von panbakteriellen PCRs bereits bekannt [48] und limitiert die Aussagekraft von auf panfungalen PCRs basierenden Untersuchungen von mutmaßlich stark kontaminierten Gewebeproben.
Die wenigen korrekten Ergebnisse in den Sanger-Sequenzierungen nach panfungaler PCR wurden bei Proben festgestellt, die erstens jünger als zehn Jahre waren und zweitens keine Hinweise auf relevante Inhibition zeigten. Selbst moderate Inhibition verhinderte bereits Amplifikation und Sequenzierung der mikroskopisch identifizierten Pathogene, so dass der aus anderen Untersuchungen [5] bekannte Einfluss von Probenqualität und Probenalter auf die Ergebnisse der molekularen Diagnostik hier bestätigt werden konnte. Hierbei ist anzumerken, dass der Einfluss des Probenalters auf die Qualität der DNA in den Proben nicht unmittelbar an den semiquantitativen Ergebnissen der humanen 18S rRNA Gen-PCR abgelesen werden konnte (Daten nicht gezeigt), was der Vielfalt an unterschiedlichen Probenmaterialien und Inhibitionsgraden geschuldet ist.
Herausforderung Kontaminationsgrad – auch ein Problem für NGS
Der zu erwartende hohe Kontaminationsgrad, den die panfungalen PCRs eindrucksvoll bestätigten, machte die untersuchten Proben auch zu einer besonderen Herausforderung für die NGS-Analytik. Da im Rahmen der NGS-Untersuchung DNA--Fragmente völlig unspezifisch analysiert werden, besteht die Herausforderung bei der Auswertung in der Abgrenzung von nachgewiesenen Kontaminanten von ätiologisch relevanten Krankheitserregern. Die histologischen Ergebnisse lieferten hierzu zwar einen Anhalt, nicht jedoch eine ätiologische Klärung auf Speziesebene.
Um diesem Problem Herr zu werden, wurden für jede mit NGS identifizierte und für ätiologisch relevant erachtete Spezies jeweils Mittelwert und Standardabweichung der prozentualen Anteile der zugeordneten Sequenzfragmente (Reads) in den untersuchten Proben bestimmt. Dann wurde untersucht, ab welcher Abweichung vom Mittelwert eine Übereinstimmung mit dem histologischen Ergebnis auftrat.
Jenseits der vierten Standardabweichung wurden keine Prozentwerte mehr errechnet. Belastbare Übereinstimmungen zwischen Histologie und NGS-Resultaten fanden sich lediglich für Prozentwerte oberhalb der dritten Standardabweichung in Relation zur Gesamtzahl der pilzspezifischen Reads. Hier fanden sich in drei von sechs Fällen mit Prozentwerten oberhalb der dritten Standardabweichung eindeutige Übereinstimmungen mit der Histologie, in weiteren zwei Fällen fand sich zumindest jeweils eine Übereinstimmung unter mehreren in dieser Häufigkeit nachgewiesenen Spezies. Bei der Analyse der Prozentwerte in Relation zur Gesamtheit der Reads in der Probe war die Übereinstimmung etwas abweichend, was sich aus den zum Teil erheblichen Unterschieden in den Anteilen der zuordenbaren Reads sowie der eukaryotischen, bakteriellen und viralen Reads erklärt. Für Proben, bei denen keine der untersuchten Spezies die dritte Standardabweichung erreichte, konnte keine zuverlässige Zuordnung durchgeführt werden.
Der Ansatz NGS-Ergebnisse aus nicht-sterilen Proben mit denen einer gesunden Population zu vergleichen um ätiologische Relevanz zu definieren ist nicht neu. Ein Vergleich mit Negativkontrollproben, der in diesem Fall auf einem Subtraktionsverfahren spezifischer Reads beruhte, wurde auch von anderen Autoren als Verfahren zur Identifikation von mutmaßlich ätiologisch relevanten Erregern vorgeschlagen. Auf diese Weise gelang der Nachweis von shigatoxinproduzierenden Escherichia coli in etwa 67 % der Stuhlproben von Ausbruchspatienten [15].
Schlussfolgerung und „optimistischer Blick in die Kristallkugel“
Wenngleich ein wegweisender diagnostischer Hinweis in fünf von 17 Proben (29,4 %) noch immer ein bescheidenes Ergebnis darstellt, muss dieses Resultat in Relation zur Komplexität des Probenmaterials gewertet werden. Im Vergleich dazu gelang durch das molekulare Goldstandardverfahren der panfungalen PCRs mit anschließender Sanger-Sequenzierung ein schlüssiger Erregernachweis sogar nur in zwei von 17 Proben (11,8 %) und selbst dies nur in drei von zehn PCR-Reaktionen, die für jene zwei Proben durchgeführt wurden. Demgegenüber erfolgte mittels NGS-Analytik in Ergänzung zur auch mittels panfungaler PCR erfolgreichen Detektion von Histoplasma capsulatum und Madurella mycetomatis auch die Bestätigung von Infektionen durch Rhizopus spp., Bipolaris spp., Cladophialophora spp. und Fusarium spp. Insbesondere bei der Zuordung auf Genus- und Speziesebene kann die Histologie, wie in der hier geschilderten Studie, im Rahmen der Differenzialdiagnostik invasiver Mykosen oft nur eingeschränkt weiterhelfen [35, 41].
Somit gelang es mittels NGS-Analytik die molekulare Erregerdiskriminierung im Vergleich zur stark kontaminationsempfindlichen panfungalen PCR mit Sanger-Sequenzierung erheblich zu verbessern. Diese Beurteilung darf nicht außeracht lassen, dass die auf prozentualer Betrachtung spezifischer Reads basierende Diskriminierung von Kontaminanten und mutmaßlich ätiologisch relevanten Erregern bisher in keiner Weise standardisiert ist und weiterer Evaluation bedarf. Unter anderem wird beispielsweise die Wahl der Zahl der Negativkontrollproben, die in die Berechnung des Mittelwerts der prozentualen Verteilung der Reads eingeht, notwendigerweise Einfluss auf die Größe der Standardabweichung und damit die potenzielle Zuordnung einer ätiologischen Relevanz haben. Auch hier ist weitere Standardisierung unabdingbar. Unter dieser Perspektive können die hier vorgestellten Ergebnisse lediglich als hypothesenbildend betrachtet werden. Weitere Studien sind notwendig um Standards für die medizinische Interpretation des NGS-gestützten Erregernachweises direkt aus Probenmaterial zu definieren. Dies gilt umso mehr für stark kontaminationsanfällige Probenmaterialien wie formalinfixierte, paraffinierte Gewebeproben.
Die ausstehende Standardisierung soll jedoch keinesfalls den Wert von NGS für die Erregerdiagnostik bei ätiologisch unklaren Krankheitsbildern [14] mindern, zumal in der hier vorgestellten Studie – für eine Evaluation im Grenzbereich der Leistungsfähigkeit der Technik – extrem kompliziertes Probenmaterial gewählt wurde. Gerade für den Sanitätsdienst der Bundeswehr kann die Option der NGS-Technik nach erfolgter Standardisierung und Festlegung von interpretativen Richtlinien in Auslandseinsätzen richtungsweisend sein um sanitätsdienstlichem Personal bei der Konfrontation mit im Heimatland seltenen Infektionskrankheiten diagnostische Hilfestellungen zu liefern. Auf weitere potenzielle Perspektiven zukünftiger Nutzung der NGS-Technologie für wehrmedizinische Fragestellungen wurde andernorts ausführlich hingewiesen [49 - 52, Georgi & Antwerpen, 2014; Steinestel et al., 2015; Vette, 2015; Zautner et al., 2015].
Kernaussagen
- Der Nachweis von Erregersequenzen mittels NGS-Analytik bietet sich als zukunftsweisendes Verfahren mit großer analytischer Tiefe auch und insbesondere für den Einsatz im Sanitätsdienst an.
- Ein großer Vorteil der NGS-basierten Diagnostik aus nach Möglichkeit steril zu gewinnendem Primärmaterial aus primär sterilen Kompartimenten des Körpers besteht darin, dass sie anders als die meisten molekularen Diagnostikverfahren keinen spezifischen Verdacht voraussetzt.
- Hauptnachteil der erregerunspezifischen NGS-Analytik bleibt neben einer eingeschränkten Sensitivität die hohe Kontaminationsanfälligkeit, die sich insbesondere bei primär nicht-kontaminationsgeschützten Materialien besonders stark bemerkbar macht.
- Im Anschluss an die geschilderte „Proof-of-Principle“-Untersuchung sollten Validierungen mit größeren Probenzahlen erfolgen, um belastbare Standards für die Abgrenzung von ätiologisch relevanten Erregernachweisen vom Nachweis von Nukleinsäurekontaminationen auch aus schwierigem aber im diagnostischem Alltag immer wieder vorkommendem Probenmaterial zu definieren.
- Durch eine zukünftige Ausbringung von NGS-Technologie in sanitätsdienstlichen diagnostischen Einrichtungen kann die weitere Evaluierung und Standardisierung dieser zukunftsweisenden diagnostischen Technologie auch für die Routinediagnostik befördert werden.
Interessenkonflikt
Der Autor erklärt, dass kein Interessenkonflikt im Sinne der Richtlinien des International Committee of Medical Journal Editors besteht.
Literatur
- Mauch H: Einfluss von Lagerung und Transport auf das Überleben von Infektionserregern. Bedeutung für eine schnelle mikrobiologische Diagnostik, die Ökonomie und den Patienten. Mikrobiologe 2008; 18: 200 - 204.
- Hagen RM, Gauthier YP, Sprague LD, Vidal DR, Zysk G, Finke EJ, Neubauer H: Strategies for PCR based detection of Burkholderia pseudomallei DNA in paraffin wax embedded tissues. Mol Pathol 2002; 55: 398 - 400.
- Quach N, Goodman MF, Shibata D: In vitro mutation artifacts after formalin fixation and error prone translesion synthesis during PCR. BMC Clin Pathol 2004; 4: 1 - 5.
- Lu K, Ye W, Zhou L, Collins LB, Chen X, Gold A, Ball LM, Swenberg JA: Structural characterization of formaldehyde-induced cross-links between amino acids and desoxynucleotides and their oligomers. J Am Chem Soc 2010; 132: 3388 - 3399.
- Frickmann H, Tenner-Racz K, Eggert P, Schwarz NG, Poppert S, Tannich E, Hagen RM: Influence of parasite density and sample storage time on the reliability of Entamoeba histolytica-specific PCR from formalin-fixed and paraffin-embedded tissues. Diagn Mol Pathol 2013; 22: 236 - 244.
- Obersteller S, Neubauer H, Hagen RM, Frickmann H: Comparison of Five Commercial Nucleic Acid Extraction Kits for the PCR-based Detection of Burkholderia Pseudomallei DNA in Formalin-Fixed, Paraffin-Embedded Tissues. Eur J Microbiol Immunol (Bp) 2016; 6: 244 - 252.
- Oosterheert JJ, van Loon AM, Schuurman R et al.: Impact of rapid detection of viral and atypical bacterial pathogens by real-time polymerase chain reaction for patients with lower respiratory tract infection. Clin Infect Dis 2005; 41: 1438 - 1444.
- Fischer M, Hagen RM, Wiemer D, Priesnitz S: Perspektiven moderner PCR-Diagnostik in der Tropenmedizin. Wehrmedizin und Wehrpharmazie 2010; 3: (http://www.wehrmed.de/article/1523--perspektiven-moderner-pcr-diagnostik-in-der-tropenmedizin.html).
- Frickmann H, Warnke P, Frey C et al.: Surveillance of Food- and Smear-Transmitted Pathogens in European Soldiers with Diarrhea on Deployment in the Tropics: Experience from the European Union Training Mission (EUTM) Mali. BioMed Res Int 2015a; 2015: 573904.
- Karger BL, Guttman A: DNA sequencing by capillary electrophoresis. Electrophoresis 2009; 30: S196 - S202.
- Reischl U, Drosten C, Geißdörfer W et al: MiQ 1. Mikrobiologisch-infektiologische Qualitätsstandards. Nukleinsäure-Amplifikationstechniken (NAT). 3. Auflage. Urban & Fischer, München & Jena; 2011: 51 - 53.
- Habib G, Lancellotti P, Antunes MJ et al.; Document Reviewers, Erol Ç, Nihoyannopoulos P et al.: 2015 ESC Guidelines for the management of infective endocarditis: The Task Force for the Management of Infective Endocarditis of the European Society of Cardiology (ESC). Endorsed by: European Association for Cardio-Thoracic Surgery (EACTS), the European Association of Nuclear Medicine (EANM). Eur Heart J 2015; 36: 3075 - 3128.
- Frickmann H, Dekker D, Schwarz NG et al.: 16S rRNA Gene Sequence-Based Identification of Bacteria in Automatically Incubated Blood Culture Materials from Tropical Sub-Saharan Africa. PLoS One 2015b; 10: e0135923.
- Bhatt AS, Freeman SS, Herrera AF et al.: Sequence-based discovery of Bradyrhizobium enterica in cord colitis syndrome. N Engl J Med 2013; 369: 517 - 528.
- Loman NJ, Constantinidou C, Christner M et al.: A culture-independent sequence-based metagenomics approach to the investigation of an outbreak of shiga-toxigenic Escherichia coli O104:H4. JAMA 2013; 309: 1502 - 1510.
- Carrick DM, Mehaffey MG, Sachs MC et al.: Robustness of next generation sequencing in older formalin-fixed, paraffin-embedded tissue. PLoS One 2015; 10: e0127353.
- Ambulos NP Jr, Schumaker LM, Mathias TJ et al.: Next-Generation Sequencing-Based HPV Genotyping Assay Validated in Formalin-Fixed, Paraffin-Embedded Oropharyngeal and Cervical Cancer Specimens. J Biomol Tech 2016; 27: 46 - 52.
- Frickmann H, Loderstaedt U, Racz P et al.: Detection of tropical fungi in formalin-fixed, paraffin-embedded tissue: still an indication for microscopy in times of sequence-based diagnosis? Biomed Res Int 2015; 2015: 938721.
- Drmanac S, Kita D, Labat I et al.: Accurate sequencing by hybridization for DNA diagnostics and individual genomics. Nat Biotechnol 1998; 16: 54 - 58.
- Ronaghi M, Pettersson B, Uhlén M, Nyrén P: PCR-introduced loop structure as primer in DNA sequencing. Biotechniques 1998; 25: 876 - 884.
- Margulies M, Egholm M, Altman WE et al.: Genome sequencing in microfabricated high-density picolitre reactors. Nature 2005; 437: 376 - 380.
- Shendure J, Porreca GJ, Reppas NB et al.: Accurate multiplex polony sequencing of an evolved bacterial genome. Science 2005; 309: 1728 - 1732.
- Branton D, Deamer DW, Marziali A et al.: The potential and challenges of nanopore sequencing. Nat Biotechnol 2008; 26: 1146 - 1153.
- Harris TD, Buzby PR, Babcock H et al.: Single-molecule DNA sequencing of a viral genome. Science 2008; 320: 106 - 109.
- Turcatti G, Romieu A, Fedurco M, Tairi AP: A new class of cleavable fluorescent nucleotides: synthesis and optimization as reversible terminators for DNA sequencing by synthesis. Nucleic Acids Res 2008; 36: e25.
- Koboldt DC, Steinberg KM, Larson DE, Wilson RK, Mardis E: The next generation sequencing revolution and its impact on genomics. Cell 2013; 155: 27 - 38.
- Hasman H, Saputra D, Sicheritz-Ponten T et al.: Rapid whole-genome sequencing for detection and characterization of microorganisms directly from clinical samples. J Clin Microbiol 2014; 52: 139 - 146.
- Kommedal Ø, Wilhelmsen MT, Skrede S et al.: Massive parallel sequencing provides new perspectives on bacterial brain abscesses. J Clin Microbiol 2014; 52: 1990 - 1997.
- Wilson MR, Naccache SN, Samayoa E et al.: Actionable diagnosis of neuroleptospirosis by next-generation sequencing. N Engl J Med 2014; 370: 2234 - 2244.
- Fischer N, Indenbirken D, Meyer T et al.: Evaluation of unbiased next-generation sequencing of RNA (RNA-seq) as a diagnostic method in influenza virus-positive respiratory samples. J Clin Microbiol 2015; 53: 2238 - 2250.
Verfasser
Oberfeldarzt Priv.-Doz. Dr. Hagen Frickmann
Fachbereich Tropenmedizin am Bernhard-Nocht-Institut,
Bundeswehrkrankenhaus Hamburg
Bernhard-Nocht-Straße 74, 20359 Hamburg
E-Mail: Frickmann@bni-hamburg.de
Kurzfassung eines der beiden Siegerbeiträge im Wettbewerb um den Paul-Schürmann-Preis 2016 der Deutschen Gesellschaft für Wehrmedizin und Wehrpharmazie e. V., der auf dem 47. Jahreskongress der Gesellschaft in Neu-Ulm am 7. Oktober 2016 vergeben wurde.
Datum: 12.04.2017
Quelle: Wehrmedizinische Monatsschrift 2017/2-3